Outline
概要
近年の研究から、腸内細菌は肥満や炎症、感染症やがんなど、今まで考えられていた以上に私たちの健康に大きな影響を与えていることがわかってきました。私たちは2015年度より健康な人を対象とした腸内細菌叢データベース構築のための研究に着手してきましたが、この度、世界最大規模の腸内細菌叢データベースとしてNIBN JMD (Japan Microbiome Database) を構築し、一部のデータを公開しました。
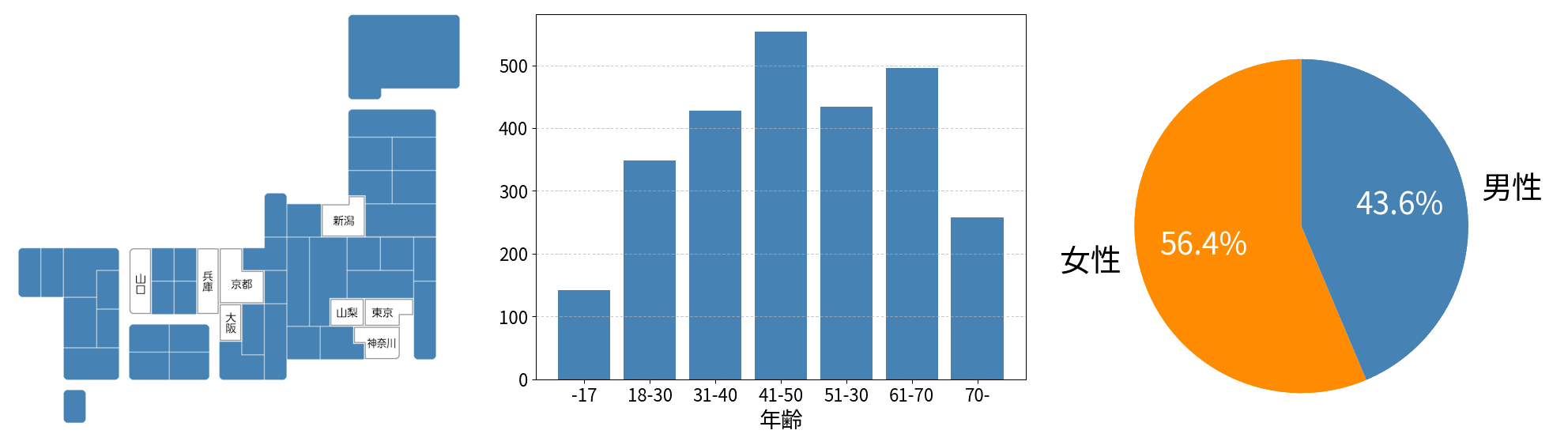
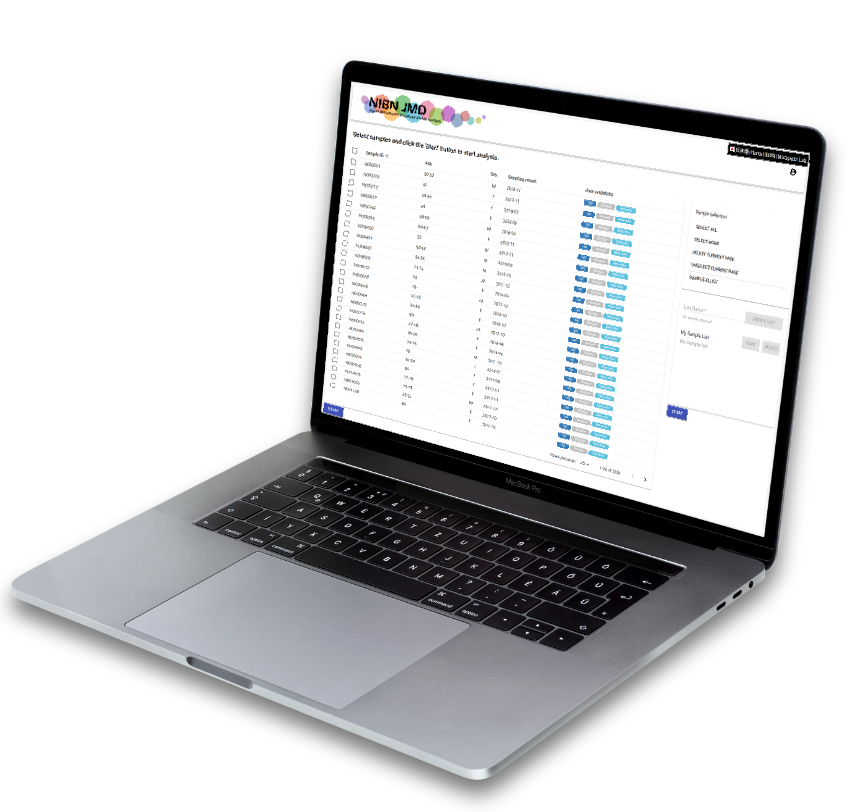
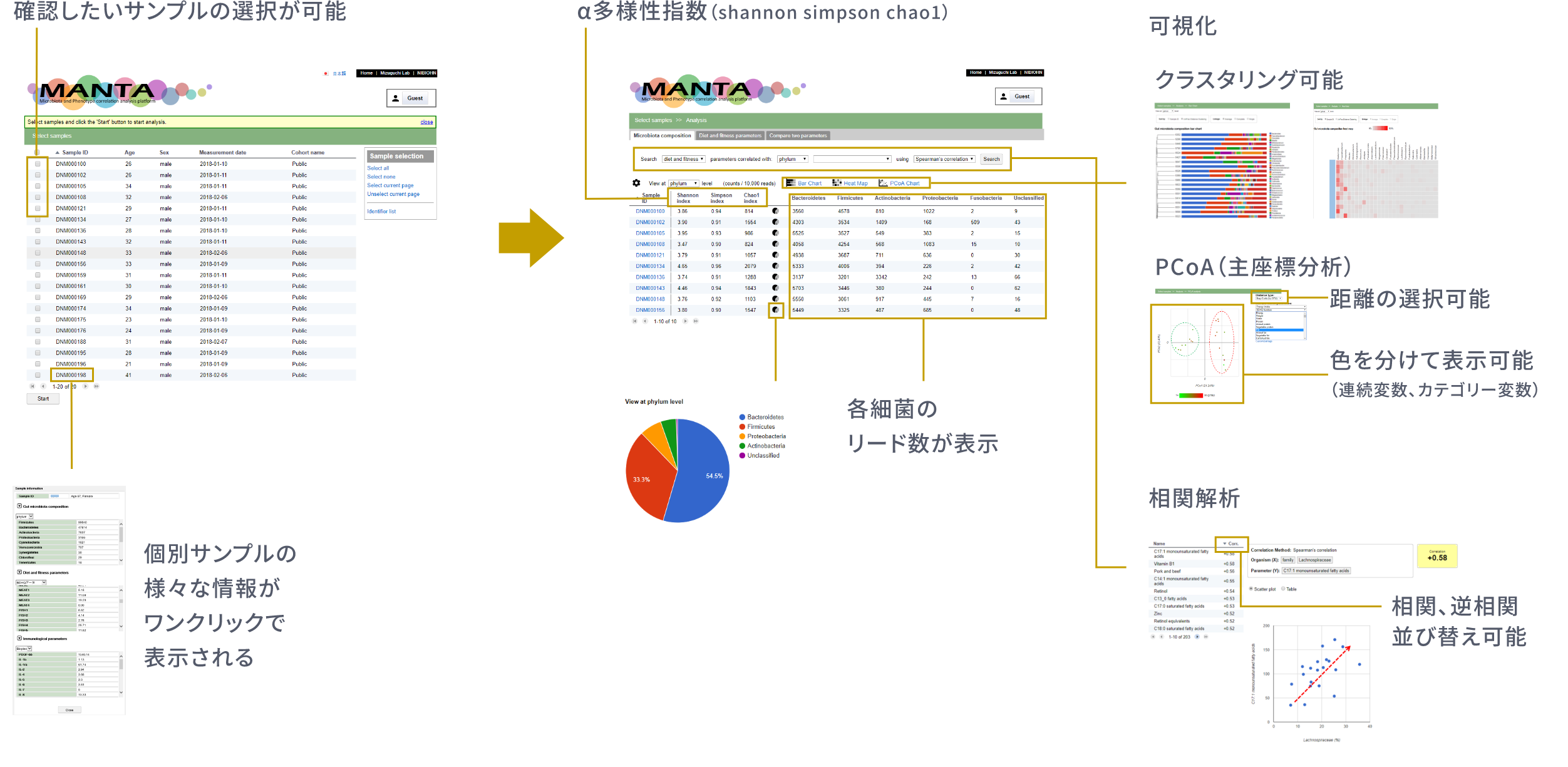
NIBN JMD には、16S rRNA遺伝子アンプリコンシーケンシングとショットガンメタゲノムシーケンシングの腸内細菌叢データに加え、食事や摂取栄養素、身体活動などの生活習慣に関する様々なデータ (表現型メタデータ) の項目情報が格納されています。また一部の参加者については、経時的な測定データや、口腔内や皮膚などの細菌叢のデータを格納しています。さらには、可視化や関連解析を可能とする独自に開発した統合解析プラットフォームMANTAを用いた解析も可能となっています。
NIBN JMDのデータは、日本人の腸内細菌叢の特徴の理解や、様々な健康状態や疾患と腸内細菌との関わりを解明する研究など、様々な研究に利用されることが期待されます。
腸内細菌

食事
運動